
[
 | E-CELL2 User's Manual | 2章 チュートリアル [ |
|
1 E-CELL2の特徴 2 E-CELL2.25のデモンストレーション 3 E-CELL2のルール作成  3.1 スプレッドシート作成 3.1 スプレッドシート作成  3.2 ルールファイルとルール・インターメディエイト・ファイル作成 3.2 ルールファイルとルール・インターメディエイト・ファイル作成 4 E-CELL2のユーザー定義Reactorの作成  4.1 Reactorの概略 4.1 Reactorの概略  4.2 Reactorの作成手順 4.2 Reactorの作成手順 5 E-CELL2の操作方法 |
この章では、E-CELL2.25の特徴と操作方法について、概略を説明して いきます。E-CELLの理論的背景についての詳細は、 the E-CELL.Org(http://www.e-cell.org)で 配布(予定)のチュートリアルを参照して下さい。
細胞をモデル化するためには、構成要素の抽象化が必要になってきます。 E-CELLでは、細胞の構造と、細胞内の化学反応を表現するために、 "Substance-Reactor Model"を採用しています。 このモデルには、以下のような特徴があります。
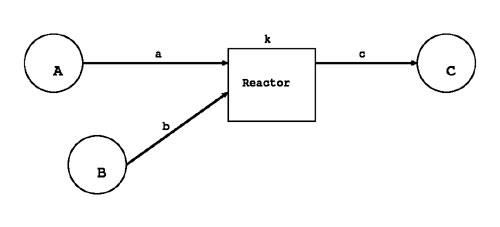
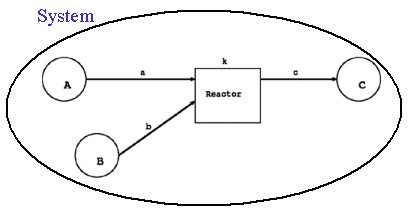
ここで注意しなければならないのは、物質(Substance)という概念が、 通常の化学分子、イオン、ラジカルのみならず、浸透圧や細胞の体積 といった、任意の物理量まで含んでいることです。但し、負の値は表現できません。
E-CELLでは、細胞内が均一であることを仮定しています。従って、拡散を 取り扱うには、物質の量が異なる、複数のシステムを用いる必要があります。
E-CELLでは細胞内の様々な現象をシミュレーションするために、 酵素反応のような反応速度(流束)に基づく反応と化学平衡のような一瞬で起こる反応を混在させることができます。
まず、流束に基づく(微分方程式として解ける)化学反応は、E-CELL内部では 次のようにして扱われます。
ソフトウェア工学的な見地からは、E-CELL2は以下のような特徴を 持っています。まず、C++言語で記述されている部分がどうなっているかを 見てみましょう。この部分はE-CELL2の心臓にあたり、E-CELL2が 細胞をどう表現しているかを示す、世界観(Ontology)に相当します。構成部品は オブジェクト指向モデリングの用語ではクラスと呼ばれ、各々のクラスが 作用しあって、シミュレーションを行うための計算機構が実現されます。
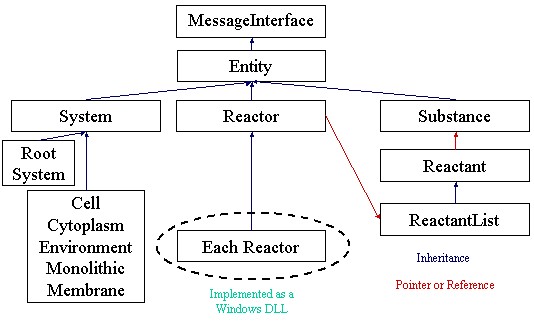
Linux用に開発された、E-CELL1から変更された点を挙げておきましょう。
これらの処理は、Linux上で動作していたE-CELL1の移植性を高めるために 行われました。一方、GUI (Graphic User Interface)や、E-CELL2の操作を 自動化するための、スクリプトファイルを解釈する部分や、モデリング・ランチャーは、 Java言語で記述されています。
E-CELL2には、対話的に操作可能なGUI版と、コマンドラインから実行 可能なバッチ版があります。2章では、GUI版についてのみ説明を行います。バッチ版の説明は 3章2.9節"E-CELL2 バッチ版の使用とLoggerの説明" にあります。GUI版とバッチ版の精度は、それぞれ64ビットと80ビットであることに注意して ください。GUI版で精度が低くなるのは、JavaのJava Native Interfaceの機構に数値計算の 精度の制約が存在するためです。
それでは、具体的なモデルを用いて、E-CELL2を動かしてみましょう。 この節では、ヒト赤血球の代謝モデルを用いて、E-CELL2を使ってみます。 何故赤血球がシミュレーションの対象として適しているのでしょうか? それには、以下のような理由が挙げられます。
赤血球の主な代謝系は、解糖系、ペントースリン酸経路、核酸合成経路 より構成されています。この他に、酸素運搬を行うヘモグロビンが大量に 存在していて、赤血球は、これらの代謝系による自己調節機能を持った、 大量のヘモグロビンが詰まった袋になぞらえることができます。 今回のモデルでは、代謝経路の再構成を目的としていますが、将来的には、 ヘモグロビンの運搬も別のモデルで表現され得ると考えられます。
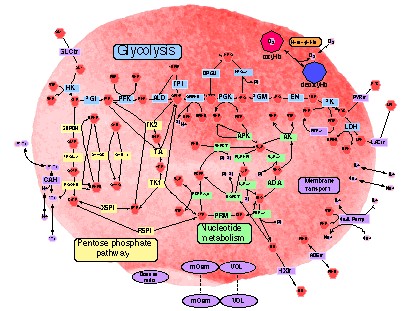
このヒト赤血球細胞の代謝モデルは、慶應義塾大学の中山講師、冨田教授が 中心になって構築されました。より詳細な情報については、the E-CELL.Org の研究紹介ページ(http://www.e-cell.org/poster/ynakayam/ISMB99/ppframe.htm) をご覧下さい。今回のデモンストレーションでは、正常なヒト赤血球細胞の代謝モデルを 扱っていますが、遺伝子異常による貧血のシミュレーションも行われています。 興味のある方は、中山講師(ynakayam@sfc.keio.ac.jp)にメールを送って下さい。
赤血球細胞のシミュレーションを見るには、 [スタート]から[プログラム]-[E-CELL2]-[erythrocyte]を選択するか、デスクトップ上のアイコンをクリック します。
赤血球モデル用のE-CELL2が起動します。E-CELL2 は、"default.ecs"というファイルがECELL2.BATと同じディレクトリにあると、 このファイルを読んでシミュレーション開始を試みます。今回の場合、図4の赤血球モデル用に default.ecsが用意されているので、自動的にデモンストレーションが開始されます。 シミュレーションのための初期値や、パラメータを記述しているファイルは、"Erythrocyte_v236.eri" です。
".ecs"で表されるファイルは一般に、E-CELL2を自動化するための スクリプトで、この中にE-CELL2を操作するための命令が書き込まれて います。詳細は、2.3節と3.4節を参照して下さい。スクリプト実行に より、図4に示した赤血球モデルのシミュレーションが開始されます。 このスクリプトでは、500秒に達した時点で計算が停止します。
スクリプトによる停止命令前にE-CELL2を終了させるには、コントロール パネルの"Stop"ボタンを押してシミュレーションを停止し、[File]メニュー から[Quit]を選択します。
次の節では、E-CELL2の操作方法について、一通り解説されます。 操作方法を理解したら、E-CELL2を再び起動して、色々と操作して 遊んでみましょう。
具体的な説明に入る前に、シミュレーション用の簡単なモデルを 紹介しておきます。下の図は、E-CELLモデリスト達に"Toy(おもちゃ)" と呼ばれる、最も簡単なフィードバック系です。
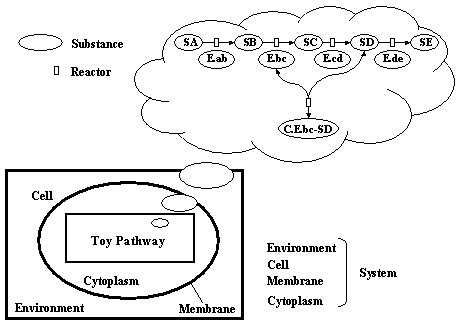
このモデルでは、5つの"S"ubstrate(基質)、 4つの"E"nzyme(酵素)、 1つの"C"omplex(複合体)が登場し、 4つのReactor(化学反応式)によりSubstrate間が結びつけられます。Reactorは、例えばMichaelis-Menten反応のような化学反応式を体現しています。 これらの反応は、培養細胞であれば培地に相当するEnvironment(環境)の中に浮かぶCell(細胞)の中に存在しています。細胞は、Membrane(膜)により 外界と内部が区別され、細胞の内部はCytoplasm(細胞質)で満たされています。
これで、化学反応のネットワークを静的なグラフとして表現できました。
しかし、実際にシミュレーションを始めるためには、個々の反応が、 どのようなパラメータ(例えば初期値や反応速度)のもとで、どのように 反応が進むか(どの化学反応式を選ぶか)ルールを決めてあげる必要が あります。
E-CELL用のモデルを作る時には、通常".er"の拡張子で識別される、ルールファイルという シミュレーションのルールをテキストエディターで記述します。それから、E-CELLに付属する ツールを使って、ルールファイルから、ルール・インターメディエイト・ ファイルという、E-CELLが読み込み可能なファイルを作成します。 ルール・インターメディエイト・ファイルは、通常".eri"の拡張子で識別されます。
E-CELLを初めて使う人が、最初からルールファイルを記述するのは 難しいので、ルールファイル作成を支援するために、スプレッド・ シートから、ルールファイルを作成するツールも提供されています。
ここではシミュレーション用スプレッド・シートを作成し、 E-CELL2のルール・インターメディエイト・ ファイルに変換する方法の概略を説明します。
E-CELL2付属のモデリングランチャを使って、ルールファイルを 作成するのに最低限必要な操作方法について説明します。
モデリングランチャを起動するには、 [スタート]から[プログラム]-[E-CELL2]-[ModelingLauncher]を選択するか、デスクトップ上のアイコンをクリック します。
E-CELL2のサンプルのルールファイルを読み込んでみましょう。 Spread sheet fileの欄のFile..ボタンをクリックし、Editを選択しファイル選択画面を開きます。 "sample.txt"を探し、このファイルを読み込んでください。
編集が終わったら、[File]メニューから、 [Save]または[Save As]を選んで、保存してください。
スプレッドシートからルールファイル、ルールファイルから ルール・インターメディエイト・ファイルを作成するための 手順を説明します。ルールファイルについての詳細は、4章を 参照してください。
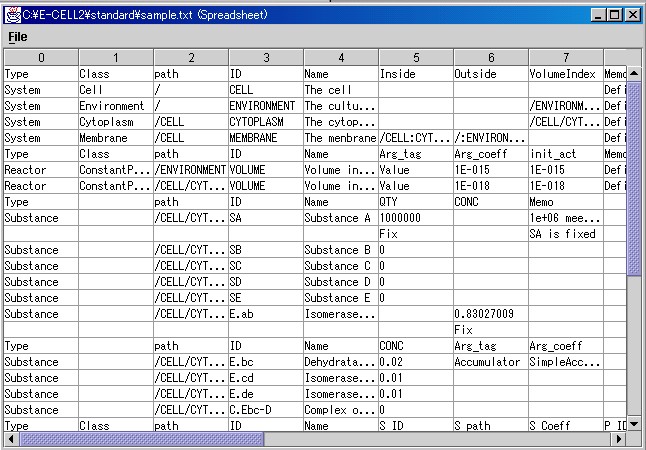
モデリングランチャーを起動して、 "sample.txt"と"toy.txt"を読み込みましょう。"toy.txt"は、 Toyモデルのスプレッド・シートが一部未完成なファイルです。 これらのファイルは、チュートリアル用に"standard"ディレクトリ に置いてあります。
E-CELLのSubstance-Reactorモデルには、 "System"、"Substance"、 "Reactor"があると既に説明しました。 スプレッドシートの一番左のカラムは、Typeと呼ばれ、各フィールドにはこれらの内のひとつが 入ります。Systemは、細胞の構造や物質の局在を示すために使用され、 代表的なものとして、"Environment(環境)"、"Cell(細胞)"、 "Membrane(膜)"、"Cytoplasm(細胞質)"があります。Systemについての詳細な説明は、4.2.2節を参照して下さい。ここでは、"Inside(内部)"、 "Outside(外部)"を区別することで、細胞等の入れ子構造が表現できることを覚えておいて下さい。
Substanceは広い意味での物質を意味します。スプレッドシートの 中には、次の表のような記述があると思います。"Toy"モデルにおけるSubstanceは、化学反応のための材料と、反応を触媒する酵素になります。
表2.1 化学反応の材料となるSubstanceの記述例
| Type | path | ID | Name | QTY |
|---|---|---|---|---|
| Substance | /CELL/CYTOPLASM | SA | Substance A | 1000 |
| FIX | ||||
| Substance | /CELL/CYTOPLASM | SB | Substance B | 0 |
| Substance | /CELL/CYTOPLASM | SC | Substance C | 0 |
PathはSubstanceが存在する場所で、UNIXのディレクトリ・パス風に 記述されます。IDは指定したSubstanceのIDで、必須項目です。Nameは指定したSubstanceの名称です。QtyとConcはどちらか片方を 記述しなければならない項目で、QtyはSubstanceの"Quantity (個数)"、Concは"Concentration (濃度)"の初期値です。Concの単位にはmol/l を使用します。QuantityやConcをシミュレーション中に変化させない場合には、直下の行に"Fix"タグ をつけて、数値を固定できます。Substanceの詳細については、4.2.3節を参照して下さい。
以下は酵素に関する記述です。
表2.2 酵素として反応を触媒するSubstanceの記述例
| Type | path | ID | Name | CONC |
|---|---|---|---|---|
| Substance | /CELL/CYTOPLASM | E.bc | Enzyme B | 0.02 |
| Substance | /CELL/CYTOPLASM | E.cd | Enzyme C | 0.01 |
| Substance | /CELL/CYTOPLASM | C.E.bc-SD | Complex of E.bc and SD | 0 |
<課題>
一方、Reactorは反応が実体化したもので、例えば以下のように 定義されます。
表2.3 Reactorの記述例
| Type | Class | path | ID | Name | S_ID | S_path | S_coeff | P_ID | P_path | P_coeff | C_ID | C_path | Arg_tag | Arg_coeff |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Reactor | MichaelisUniUniReactor | /CELL/CYTOPLASM | E.ab-0 | A->B | SA | /CELL/CYTOPLASM | 1 | SB | /CELL/CYTOPLASM | 1 | E.ab | /CELL/CYTOPLASM | KmS | 10 |
| KcF | 5 | |||||||||||||
| Reactor | MichaelisUniUniReactor | /CELL/CYTOPLASM | E.bc-0 | B->C | SB | /CELL/CYTOPLASM | 1 | SC | /CELL/CYTOPLASM | 1 | E.bc | /CELL/CYTOPLASM | KmS | 0.1 |
| KcF | 3 | |||||||||||||
| Reactor | MichaelisUniUniReactor | /CELL/CYTOPLASM | E.cd-0 | C->D | SC | /CELL/CYTOPLASM | 1 | SD | /CELL/CYTOPLASM | 1 | E.cd | /CELL/CYTOPLASM | KmS | 0.1 |
| KcF | 2 |
PathはReactorが存在する場所で、UNIXのディレクトリ・パス風に 記述されます。S_IDは"Substrate(基質)"で、化学反応の出発物質 を意味し、S_Pathはその存在する場所です。P_IDは"Product(産物)"で、 化学反応の結果生成した物質を意味し、P_Pathはその存在する場所です。C_IDは"Catalyst(触媒)"で、反応を促進あるいは抑制する化学物質 を意味し、C_Pathはその存在する場所です。Arg_tag列には、Reactorの 種類によって定義されている、定数名が入力されます。Arg_coeffには、Arg_tagに対応する定数値が入力されます。
IDが!で始まるReactorは、裏口リアクターで、微分方程式では 記述できない反応を記述するのに使用されます。"Toy"モデルでは、 "!EQ-Ebc-D"が代数式の裏口リアクターです。Ebc+SD<->C.Ebc-Dという 反応が、EbcとSDからC.Ebc-Dが生成する反応と、C.Ebc-Dの分解する 反応の可逆反応なため、微分方程式では解くことができません。 そのため、迅速平衡用の"RapidEquibriumPReactor"を利用します。
<課題> 1. ReactorのD->Eの反応について、記述を補ってください。
Reactorの詳細については、4章2.4節"スプレッドシートの記述・ Reactorパート"を参照して下さい。 ここまでの作業が終わったら、"toy.txt"を保存して、 ルールファイルを作成する作業に入りましょう。
それでは、スプレッド・シートからルールファイルを作成する 実際の操作をモデリングランチャーを使いながら説明していきましょう。
Spread sheet fileの欄の"File..."ボタンをクリックし、"Choose..."を選択しファイル選択画面を開きます。 "toy.txt"を探し、このファイルを読み込んでください。
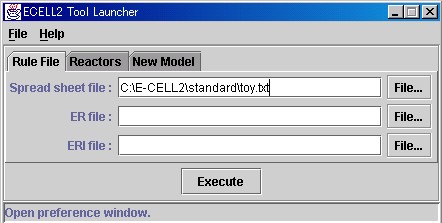
Executeボタンをクリックすると、ルールファイル"toy.er"とルール・インターメディエイト・ファイル"toy.eri"ファイルが作成されます。
ルールファイルやルール・インターメディエイト・ファイル作成に関する説明は以上です。 次のReactorを作成するステップに進みましょう。
標準リアクターにないような化学反応を記述したい場合は、あなた自身が、 E-CELL2用にReactorをデザインする必要があります。
RDファイルとはキーワードと値の組からなる行を、必要な数だけ並べて作成するものです。
RDファイルの構成は大きくリアクター定義ファイルを作成する際には以下の約束事に注意する必要があります。
RDファイルの例を以下に示します。
| v = | KcF[E][S] | |
|
||
| KmS + [S] |
MichaelisUniUniReactorについてのファイル
@CLASSNAME:MichaelisUniUniReactor
@BASECLASS: FluxReactor
@AUTHOR: E-CECLL Tutorial
@EMAIL: tutorial@e-cell.org
@DATE: 2000 12/12
%VERSION: ecs-v1, 0.1
@BRIEF_DESCRIPTION:Unireactor enzyme activity of which kinetics can be described by the Henri-Michaelis-Menten equation.
@DESCRIPTION:A reactor class for unireactant enzyme activity where kinetics can be described
by the Henri-Michaelis-Menten equation derived from rapid equilibrium assumptions.
\vspace{0.2cm}
This reactor is applicable to the following reaction sequence:
\begin{center}
$E+S \rightleftharpoons ^{k_{1}}_{k_{-1}}ES
\rightarrow^{k_{p}}E+PS
\end{center}
\vspace{0.3cm}
@EQUATION:$$v=\frac{(K_{cF}[E] [S]}{K_{mS}+[S]}$$
%SUBSTANCE:Substrate, 1, 1
%SUBSTANCE:Product, 1, 1
%SUBSTANCE:Catalyst, 1, 1
%SUBSTANCE:Effector, 0, 0
%PARAMETER: KmS, Float, mol/l, Michaelis Constant of Substrate
%PARAMETER: KcF, Float, mol/l, Catalytic Constant (Forward)
@REACT_FUNC:
Float S = substrate(0)->concentration();
Float E = catalyst(0)->quantity();
Float velocity = KcF * E * S;
Float Den = KmS + S;
velocity /= Den;
process(velocity);
以上がReactorに関する簡単な説明です。Reactorの詳細については、5章を参照してください。
簡単な例を挙げて、Reactorをどのように作成していくかを説明します。
モデリングランチャーを起動し、Reactorsのタブをクリックします。 RD fileの欄の"File.."ボタンをクリックし、"Edit..."を選択しファイル選択画面を開きます。
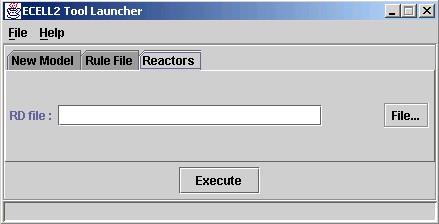
"MyMichaelisMentenUniUni.rd"というファイルを開いてみましょう。リアクター定義 ファイル(RDファイル)は、通常".rd"という拡張子を持つファイルとして作成されます。 このファイルの中には、リアクターの定義が記述されています。
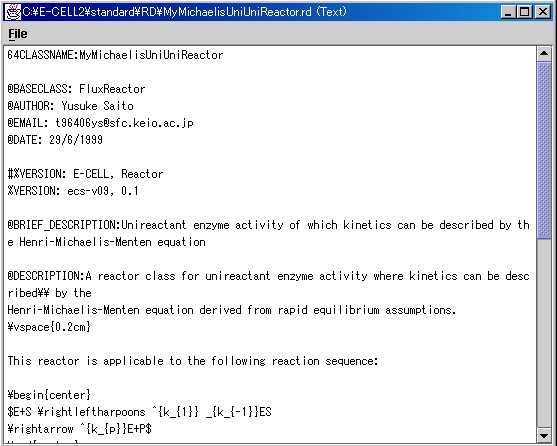
リアクター定義ファイルを作成する上でまず注意する必要があるのは、 拡張子を除いたファイル名が、そのまま@CLASSNAMEとならなけば ならない点です。このファイルでも一致させてあります。 MyMichelisMentenUniUniReactor.rdは、クラス名を除いては MichaelisMentenUniUniReactor.rdと内容が同一です。標準リアクター の定義ファイルは、標準的なインストールでは、"C:\E-CELL2\standard \RD"にあります。実際にリアクター定義ファイルを作成する際は、 これらのファイルの記述を参考にすると良いでしょう。
リアクター定義ファイルから、モデリングランチャーを利用してC++のソースコード とヘッダファイルを生成し、コンパイルを行なうことでリアクターDLLを作成します。 "Execute"ボタンを押してDLLを作成しましょう
RDファイル中のC++コードに文法的な誤りがなく、Borland C++ Compilerの設定が適切に行われていれば、DLLが作成されます。 実行後にはメッセージウィンドウにコンパイル時のメッセージが表示されます。
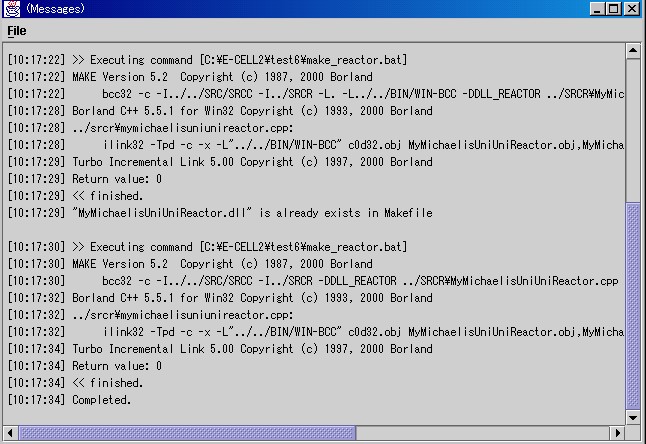
コンパイルが終わったら、実際に"MyMichaelisMentenUniUniReactor"を 使用してみましょう。モデリングランチャーを利用して"toy.txt"を開きます。
"MichaelisMentenUniUniReactor"と "MyMichaelisMentenUniUniReactor"と差し替えて、ファイルを保存 し、"Execute"ボタンを押しましょう。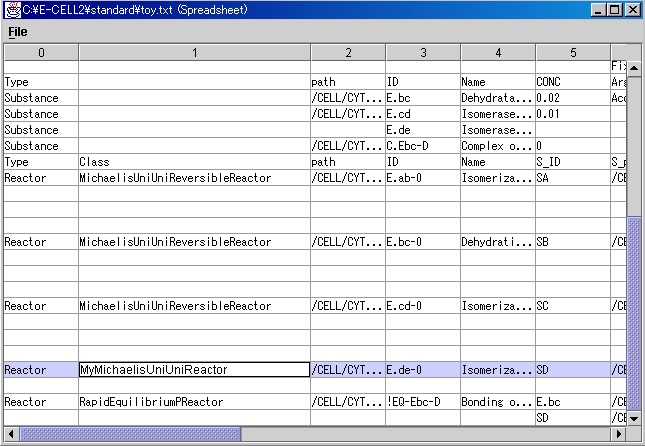
ここで、E-CELL2を起動し、"toy.eri"を[File]メニューより [Load Rule]を選んで読み込み、シミュレーションを開始して みましょう。"sample.eri"と読んで実行したときと同じ挙動で あれば、リアクター作成は成功です。
今後は、必要に応じてReactorをデザインして、独自のシミュレーション ができるように訓練を積んで行ってください。
作成したルール・インターメディエイト・ファイルとリアクターを用いて、 E-CELL2の基本的な操作を覚えましょう。E-CELL2を起動すると、 まずコントロールパネルが現れます。
まず、プルダウンメニューについて簡単に説明します。
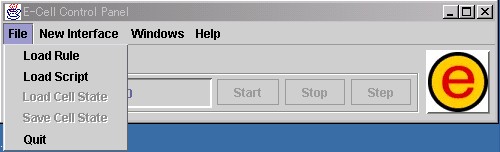
[File]メニューには、[Load Rule]、[Load Script]、[Load Cell State]、 [Save Cell State]、[Quit]の項目があります。
[Load Rule]は、ルール・インターメディエイト・ファイルを 読み込む際に選択します。
[Load Script]は、E-CELL2の操作を自動化するためのスクリプトを 読み込む際に選択します。
[Load Cell State]は、途中経過を記録したファイルを読み込んで、 シミュレーションを再開する際に選択します。
[Save Cell State]は、シミュレーションの途中経過を記録した ファイルの書き出しに使用します。
[Quit]を選択すると、E-CELL2が終了します。
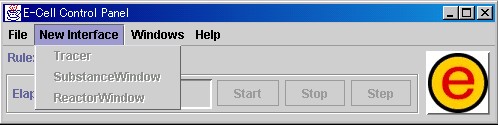
[New Interface]メニューには、[Tracer]、[SubstanceWindow]、 [ReactorWindow]の項目があります。
[Tracer]は、Substanceの量や、Reactorの活性を描画する、[Tracer] 画面を生成するために選択します。
[SubstanceWindow]は、Substanceの表示と操作を行う画面を開くために 選択します。
[ReactorWindow]は、Reactorの表示を行う画面を開くために選択します。
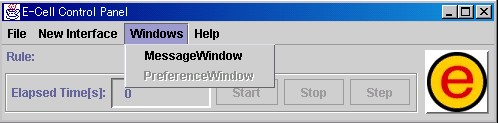
[Windows]メニューには、[MessageWindow]と[PreferenceWindow]項目 があります。
[MessageWindow]は、E-CELL2からのメッセージ画面を表示させるのに 使用します。
[PreferenceWindow]は、シミュレーションの1ステップの時間幅と、 [Tracer]画面更新のタイミングを設定する際に選択します。
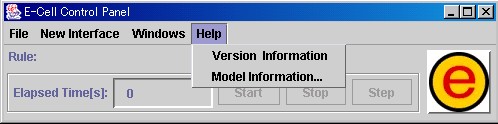
[Help]メニューには、[Version Information]項目があり、これを選択すると バージョン情報が表示されます。
以上が、メニューに関する説明です。
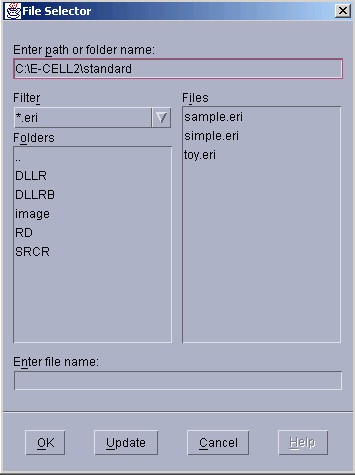
ルール・インターメディエイト・ファイルを読み込むには、[File]メニュー -[Load Rule]を選択します。ファイルを選択する画面が表示されるので、 ルール・インターメディエイト・ファイルを意味する、".eri"で終わる ファイルの名前をクリックし、 Ok (開く)"を選択しましょう。
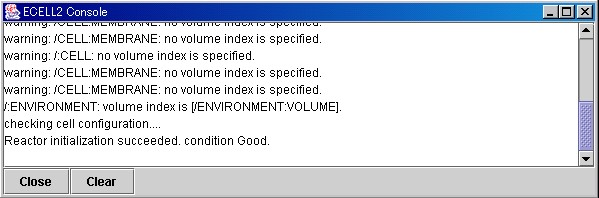
指定されたファイルが正常に読み込まれると、Message Windowに、 "Condition Good."と表示されます。
次に、[Tracer]画面を開いて、Substanceの量をグラフとして表示する ための準備をします。[New Interface]-[Tracer]を選択するとTracer が表示されます。

Tracerの左下にある"Add"ボタンを押して、[Entry Selector]を 表示します。左パネルをクリックして、Cytoplasmに位置しているSubstanceを選択してみましょう。
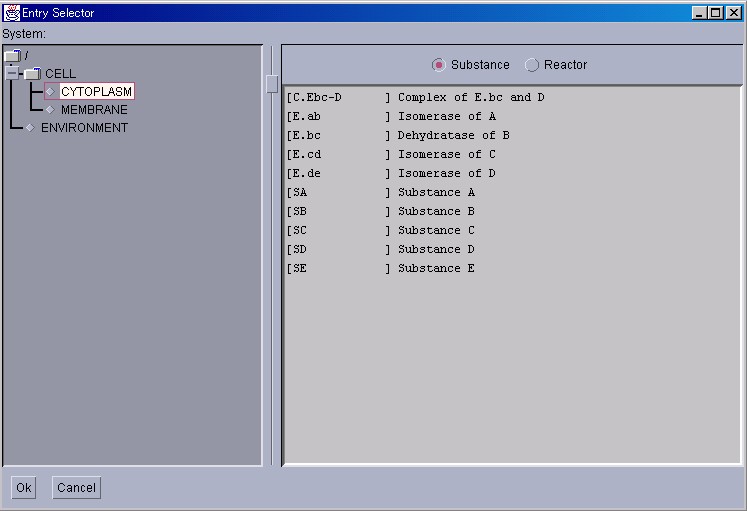
Substanceのラジオボタンをクリックすると、Substanceが右の パネルに表示されます。Reactorを表示させることも可能です。
複数のSubstanceやReactorを選択する方法を説明します。 まとめて選択するには、"Shift"キーを押しながら、マウスを 左クリックしてください。飛び飛びに選択するには、"Ctrl"キー を押しながらマウスを左クリックしてください。選択したら、 "Ok"ボタンを押します。この状態で、コントロールパネルの "Start"を押してシミュレーションを開始することができるように なります。
一枚のTracerには、8種類までのSubstanceやReactorを表示可能です。
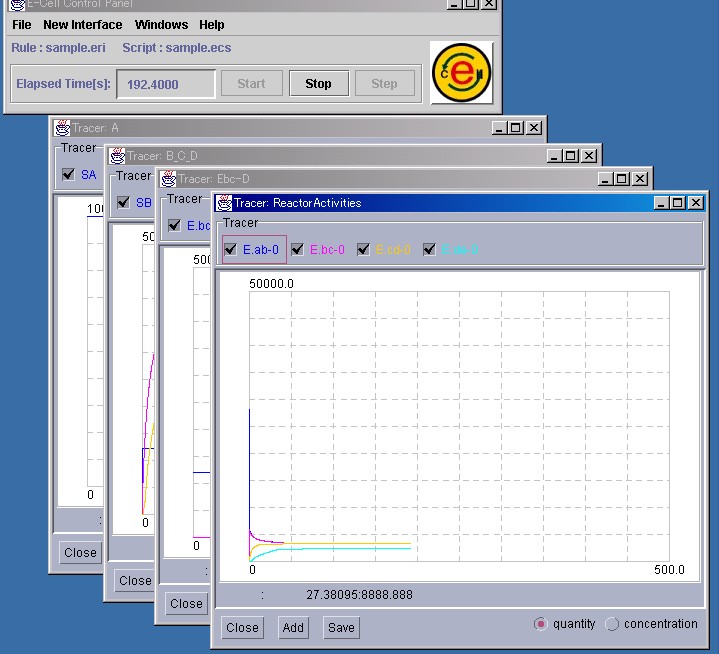
ルールに従って、Substanceの量や、Reactorの活性の変化がTracerに 表示されます。Tracerに表示されたシミュレーションの結果は、Tracerの "Save"ボタンを押すと保存できます。詳細は、3.2.7節を 参照して下さい。
Substanceの量を手動で変更して、全体の系にどのような影響が及ぶか 見ることもできます。[New Interface]メニューから、[Substance Window] を選んでみましょう。
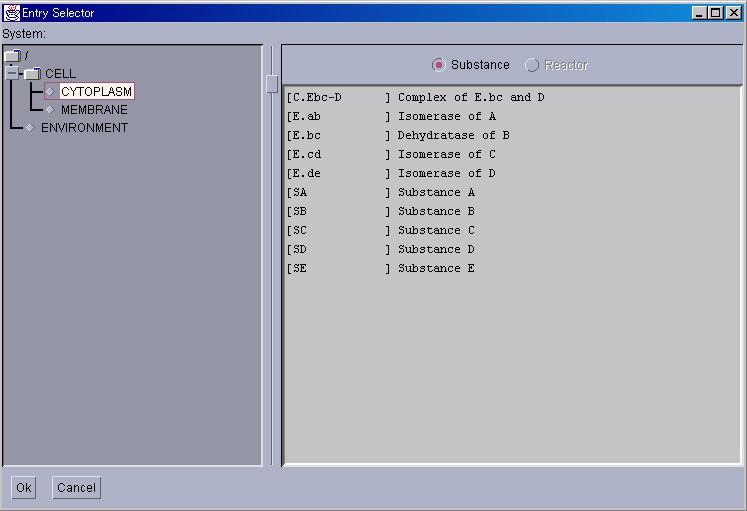
このEntry Selectorは、Substance専用なので、Reactorを選ぶことは できません。Substanceを選択し、"Ok"ボタンを押すと、選択されたSubstanceの個々の詳細情報を表示する画面が現れます。
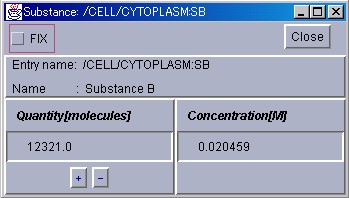
"+", "-"ボタンを押すか、"Quantity"あるいは"Concentration" テキストフィールドに値を直接入力して、注目するSubstanceの量を人為的に 増減することができます。"Fix"チェックボタンをチェックすると、 そのSubstanceの量を、計算結果を無視して一定に保つことが可能になります。SubstanceWindow の操作を終了するには、"Close"ボタンを押します。
Substanceの量を変化させて、系にどのような影響が出るか見てみましょう。
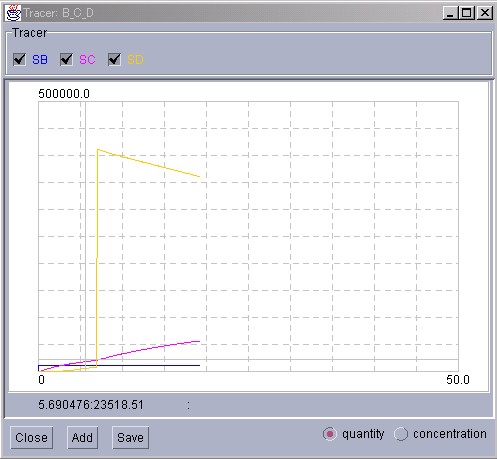
この例では、"Substance D"を急激に増加させています。このSubstanceを 増加させても、系の中の反応に補償されて、徐々に量が減っていくのが 分かります。
同様に、Reactorの活性を詳細表示することもできます。 コントロールパネルの、[New Interface]メニューから、[ReactorWindow] を選びましょう。Reactor用の[Entry Selector]が表示されます。
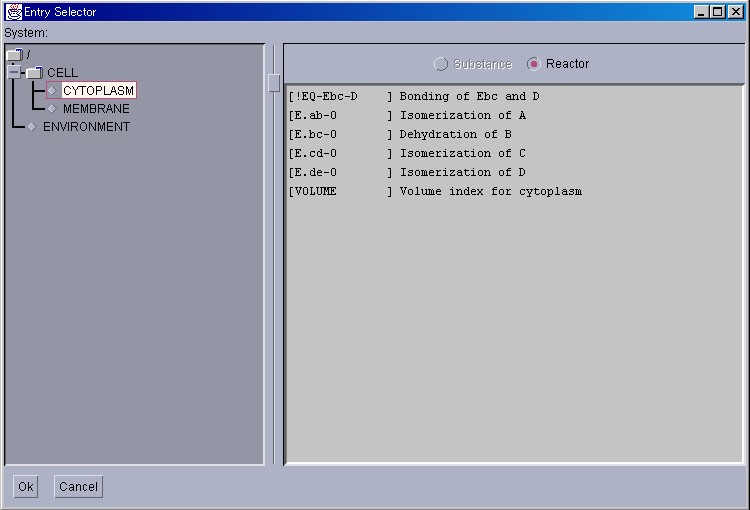
SubstanceWindowのときと同様に、左パネルに表示されるSystemを クリックして、目的のReactorを探します。Reactorを選択したら、 "Ok"ボタンをクリックしましょう。
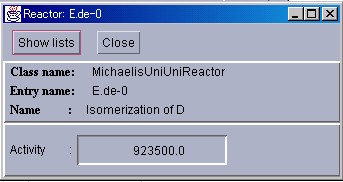
この画面では、あるReactorについて、その名前、使用している反応式の 種類、反応の活性値が表示されます。"Show lists"ボタンを押すと、更に 詳細な情報が表示され、"Close"ボタンを押すと画面が閉じます。
"Show lists"ボタンを押してみましょう。
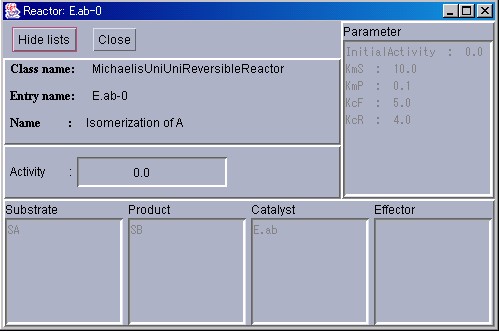
上記の情報の他に、各種パラメータの初期値、反応に関与する物質について の情報が表示されます。画面を元の大きさに戻す場合は、"Hide lists"ボタン を押し、閉じるには"Close"ボタンを押します。
次は、PreferenceWindowです。この画面を呼び出すには、コントロール パネルの[Windows]メニューから、[PreferenceWindow]を選びます。 この画面は、シミュレーションのための計算の1ステップの時間幅(単位: 秒) と、Tracerの画面更新を何ステップ毎に行うかを制御するために 用意されています。この時間幅を短く取ると、計算の精度は上がりますが、 シミュレーションにかかる時間は長くなります。逆に、時間幅を長く取ると、 シミュレーションにかかる時間は短くなりますが、計算の精度が荒くなり、 結果を信頼することができなくなってしまいます。初期設定では、 計算の1ステップが0.001秒、画面の更新頻度が100回に1回に設定されて います。
この画面はモーダルダイアログなので、"Ok"ボタンで決定するか"CANCEL"で閉じるか しないと、他の画面の操作を行うことができません。
MessageWindowには、E-CELL2からのメッセージが表示されます。このウィンドウを閉じるには、"Close"ボタンを押して下さい。 "Clear"ボタンを押すと画面上に表示されたメッセージがクリアされます。
コントロールパネルの[Help]メニューから、 [Version Information]を選択することができます。Version Information には、E-CELL2のバージョンの情報が表示されます。
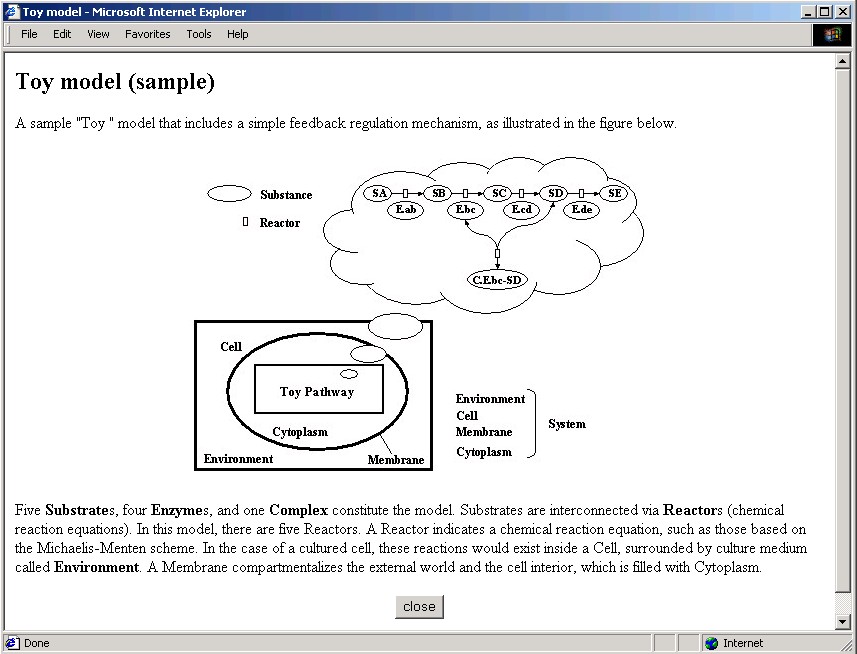
また、コントロールパネルの[Help]メニューから、 [Model Information]を選択すると、ブラウザ上にモデルの説明が表示されます。
この節の最後に、シミュレーションの状態の保存方法を説明します。 シミュレーションの途中経過を保存するには、コントロールパネル上で "Stop"ボタンを押し、[File]メニューから[Save Cell State]を選択します。
細胞モデル全体の状態を記述したファイルは、拡張子".cs"が付きます。 適当なディレクトリ上に、保存してください。この形式のファイルは、 コントロールパネルの[File]メニューから、[Load Cell State]を選択 することで読み込むことが可能で、保存された状態からシミュレーション を再開することが可能です。
大きなシミュレーションになってくると、色々な作業をいちいち手動で 行うのが大変です。そこでE-CELLでは、"スクリプト"という機構を用意 していて、スクリプトの中に記述された命令をE-CELLに読み込ませることで、 シミュレーションを自動化できます。このスクリプトファイル には、通常".ecs"という拡張子で識別されます。
ここでは、スクリプトの書き方について簡単に説明し、E-CELL1とE-CELL2 で、スクリプトファイルの形式が異なる部分についても説明します。
まず、以下のファイルを見てください(行頭の数字は行番号です。)。
01 TmpDir /temp 02 LoadRule sample.eri 03 04 NewInterface Tracer A_B_C 05 AddTrace Substance:/CELL/CYTOPLASM:SA 06 AddTrace Substance:/CELL/CYTOPLASM:SB 07 AddTrace Substance:/CELL/CYTOPLASM:SC 08 SaveAt 500 09 10 UpdateInterval 100 11 12 Run 501 13 SaveCellState sample-after500s.cs 14 Stop
1行目: 一時ファイルを保存するディレクトリを指定します。
/tempはカレントドライブの直下のtempディレクトリを意味し、インストール時に作成されます。
2行目:読み込みたいルール・インターメディエイト・ファイル
(".eri"ファイル)を指定します。
4-8行目:Tracerに関する表示
4-7行目:AddTraceはTracer内でのみ宣言される。
引数にかかれたSubstance、もしくはReactorをTracerの表示に
加える。引数は、IDだけでなくそれが存在するSystemとTypeを
含めて示す必要があります。
8行目:このTracer上に表示させた物質を、E-CECLL時間の500秒後に
保存します。Tracer内でのみ宣言されます。
9行目:NewInterfaceの終わりは、空白行が必要になります。
10行目:Tracerに表示させることを100ステップごとに行います。
12行目:501秒間シミュレーションを行います。
13行目:細胞モデルの状態を"sample-after500s.cs"というファイルに
保存します。
14行目:シミュレーションを停止します。
上記のファイルを、"toy.ecs"のような、適当な名前で保存します (ただし、US-ASCII以外の文字を使用しないで下さい。)。保存する ディレクトリは、"sample.eri"がある、"C:\E-CELL2\standard"に しましょう。これ以外のディレクトリにE-CELL2をインストールしている 場合は、適当に読み替えてください。
E-CELL2の標準リアクターモデル版を起動しましょう。起動したら、 コントロールパネルの[File]メニューから、[Load Script]を選択し、 作成した".ecs"ファイルを読み込みましょう。
それから、E-CELL2から実装された"Logger"という機構についても紹介して おきます。Loggerは、細胞の状態をシミュレーション中に随時記録する 機構です。細胞の状態をスナップショットで保存するよりも、先進的で、 スクリプトファイルに以下のような記述を追加することで利用できます。
NewInterface Logger test-log AddTrace Substance:/CELL/CYTOPLASM:SA AddTrace Substance:/CELL/CYTOPLASM:SB AddTrace Substance:/CELL/CYTOPLASM:SC AddTrace Substance:/CELL/CYTOPLASM:SD AddTrace Substance:/CELL/CYTOPLASM:E.bc AddTrace Substance:/CELL/CYTOPLASM:C.Ebc-D AddTrace Reactor:/CELL/CYTOPLASM:E.ab-0 AddTrace Reactor:/CELL/CYTOPLASM:E.bc-0 AddTrace Reactor:/CELL/CYTOPLASM:E.cd-0
最初に作成した、".ecs"ファイルの9行目の後にLoggerに関する記述を 追加して、E-CELL2に読み込ませてみましょう。
以下は、Loggerの特徴です。
シミュレーションが停止したら、Loggerディレクトリ以下の".ecd"ファイル をメモ帳(notepad)などで開いてみましょう。ファイル名が個々のSubstanceやReactorを示していて、"時刻"、"瞬間数"、"平均数"、"最大数"、 "最少数"、"瞬間濃度"、"平均濃度"、"最大濃度"、"最小濃度"の情報が 記載されています。
ここまでで一通り、シミュレーションをどのような手順で進めていくかを 説明しました。個々の項目の詳細な説明については、3章以降を 参考にしてください。
 | E-CELL2 User's Manual Last Update $Date: 2002/11/25 $. Copyright: Keio University and Mitsui Knowledge Industry Co., Ltd. 2002 | 2章 チュートリアル [ |